Nature biotechnology:小RNA-seq技术用于miRNA定量分析的多中心综合评估
摘要一览
RNA-seq越来越多地用于定量分析不同样品类型中的小RNA (例如,microRNAs、piRNAs和snoRNAs ),包括分离的细胞、组织和其他生物样本。目前使用的小RNA-seq文库制备方法的准确性和重现性尚未得到系统测试。本研究报道了由九个实验室获得的结果,这些实验室独立测序了人工合成的小RNA和人血浆衍生RNA,通过对这些“ground truth”样本的检测,对商业上可获得的三种使用接头定义序列的文库制备方法,以及六种使用简并碱基接头的方法进行了系统评估,发现了实验流程和测序中的特异性偏差区别,包括降低用小RNA-seq技术来准确测量microRNAs中腺苷到肌苷编辑能力的偏差。本研究发现,这些偏差通过结合带有简并碱基接头的文库制备方法得以改善。通过不同实验室的评估结果表明,使用小RNA-seq检测样品中microRNA的相对定量是准确和可重复的。
发表期刊:Nature biotechnology 发表时间:2018-7-16 影响因子:35.724
背景介绍
RNA-seq已经广泛应用于对生物体转录组特征的分析。RNA-seq可用于测序长的RNA ,例如信使RNAs(mRNAs)和长链非编码RNAs(lncRNAs)和短RNAs,例如小的非编码RNAs,像microRNAs等。这些应用在目标RNAs的大小、所用的技术方法以及所产生的定量数据偏差方面有所不同。例如,长RNA-seq文库的制备,具有足够长的目标RNA长度,通常利用引物从RNA直接产生cDNA。相反,小RNA-seq文库的制备方法通常需要RNA连接反应或加poly-A尾巴的过程,以克服从极短(例如16-30nt) 的目标RNA序列进行逆转录和随后PCR扩增带来的问题。
存在的问题
为了从小RNA中高效并且获得一致性好的cDNA,人们基于小RNA-seq开发了多种方法,所以用于小RNA-seq的方案要比用于长RNA-seq的方案变化更大,从而产生了来自不同文库制备方案和不同实验室的更大的变化可能性。此外,越来越多的小RNA-seq被用于研究RNA浓度非常低的样品,例如外泌体等。为校正长RNA-seq数据的变化而开发的标准化方法常常不太适合小RNA-seq数据。尽管对长RNA-seq的性能特征,如再现性和定量准确性,已经进行了深入研究,但是对于小RNA-seq,仅有报道评估了单个文库制备方案的再现性。
此外,用于定量RNA序列中单核苷酸变化的不同小RNA-seq方法的性能,还没有得到系统的检验,例如那些通过microRNA (miRNA)编辑的方法等。随着小RNA-seq数据的快速积累(例如NIH的SRA数据库、胞外囊泡(EV)相关小RNA测序数据库、癌症基因组图谱(TGCA)数据库、exRNA图谱数据库等),对结果进行有意义的定量解释,尤其是跨组学的研究,将受益于对技术偏差、其对准确性的影响以及小RNA-seq重复性的系统检查。
研究内容及意义
本研究由九个实验室进行了多方案、多机构评估,使用标准化的合成参考试剂和生物衍生参考RNA来评估小RNA-seq的准确性、重现性和技术偏差。研究还评估了不同方案在表征miRNA编辑方面的性能,并确定了一种文库制备方法,该方法减少了技术偏差,提高了小RNA-seq结果的准确性和可比性,这些都为以后测序方法的校正改进提供理论依据。
研究方法
小RNA-seq
研究结果
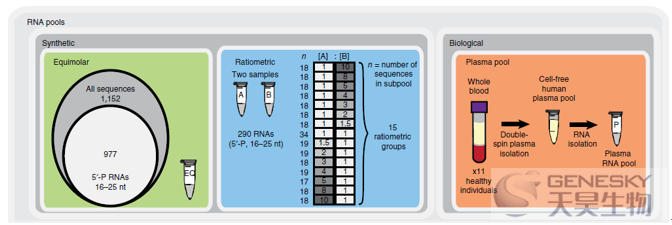
图1-实验设计I
上图显示了研究中用作普通参考样品的四个主要RNA池。样品EQ(Equlmolar)表示使用化学合成的RNA寡核苷酸制备的等摩尔池,为等摩尔数混合了1152种15-90 nt的RNA寡核苷酸,其中15-25 nt长的占到了977种。样品A和样品B表示的是使用化学合成的RNA寡核苷酸制备的不同比例的RNA池,包含了334种合成RNA,其中15-25 nt长的占290种,共分为15个亚组。这样可以分别建立绝对和相对丰度的“ground-truth”参照。样品P表示为血浆RNA库,包括来自11名健康男性的RNA,这些RNA被集中分离并分发给不同实验室。
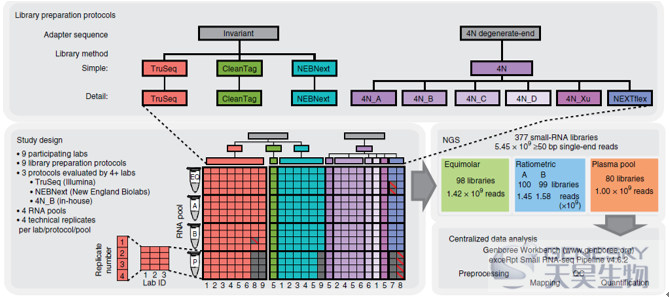
图1-实验设计II
本研究测试了九种不同的文库制备方案。测试了三个具有不变”接头“的商用试剂盒(TruSeq (Illumina)、NEBNext(New England BioLabs)、CleanTag(Trilink Biotech))和六个4N简并末端的制备方案。参考RNA池分给九个实验室,使用标准化常用TruSeq方案和至少一种额外方法进行一式四份测序。具体的建库分工情况显示在左下角彩色网格中。带有对角线红线的灰色方块表示试图进行文库制备和测序但未成功的样品。每个实验室独立进行测序。实验最终成功构建了377个小RNA文库,共获得5.45 x 10
9的reads。
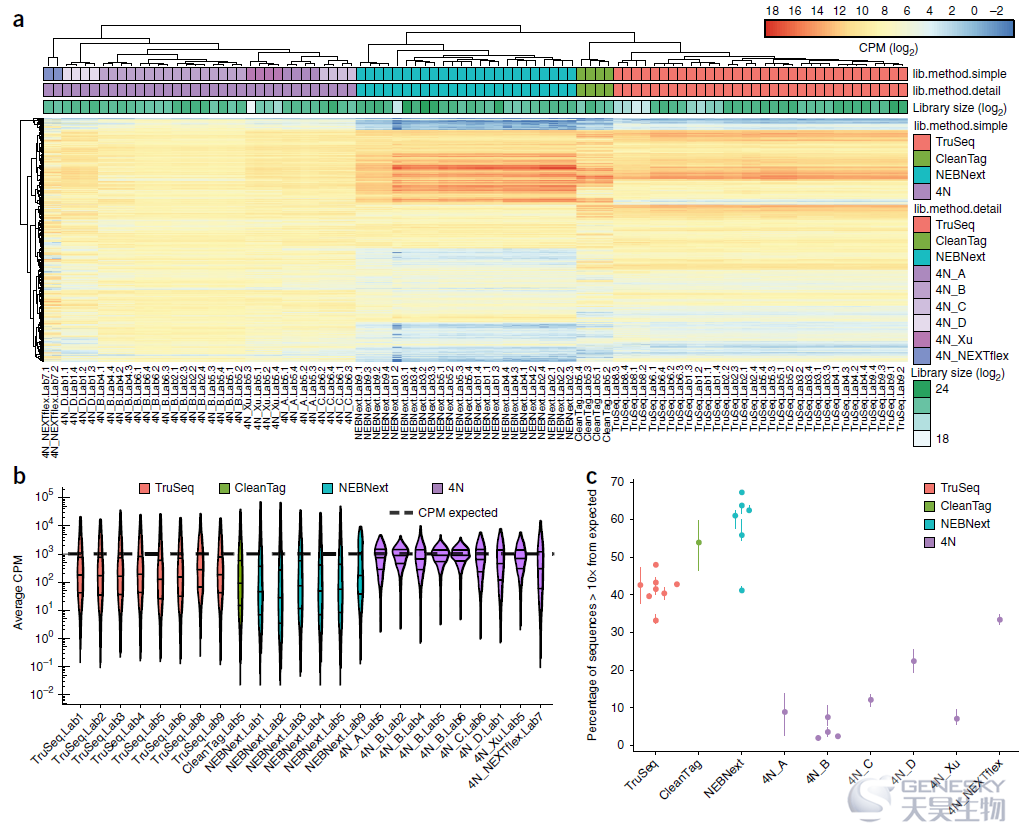
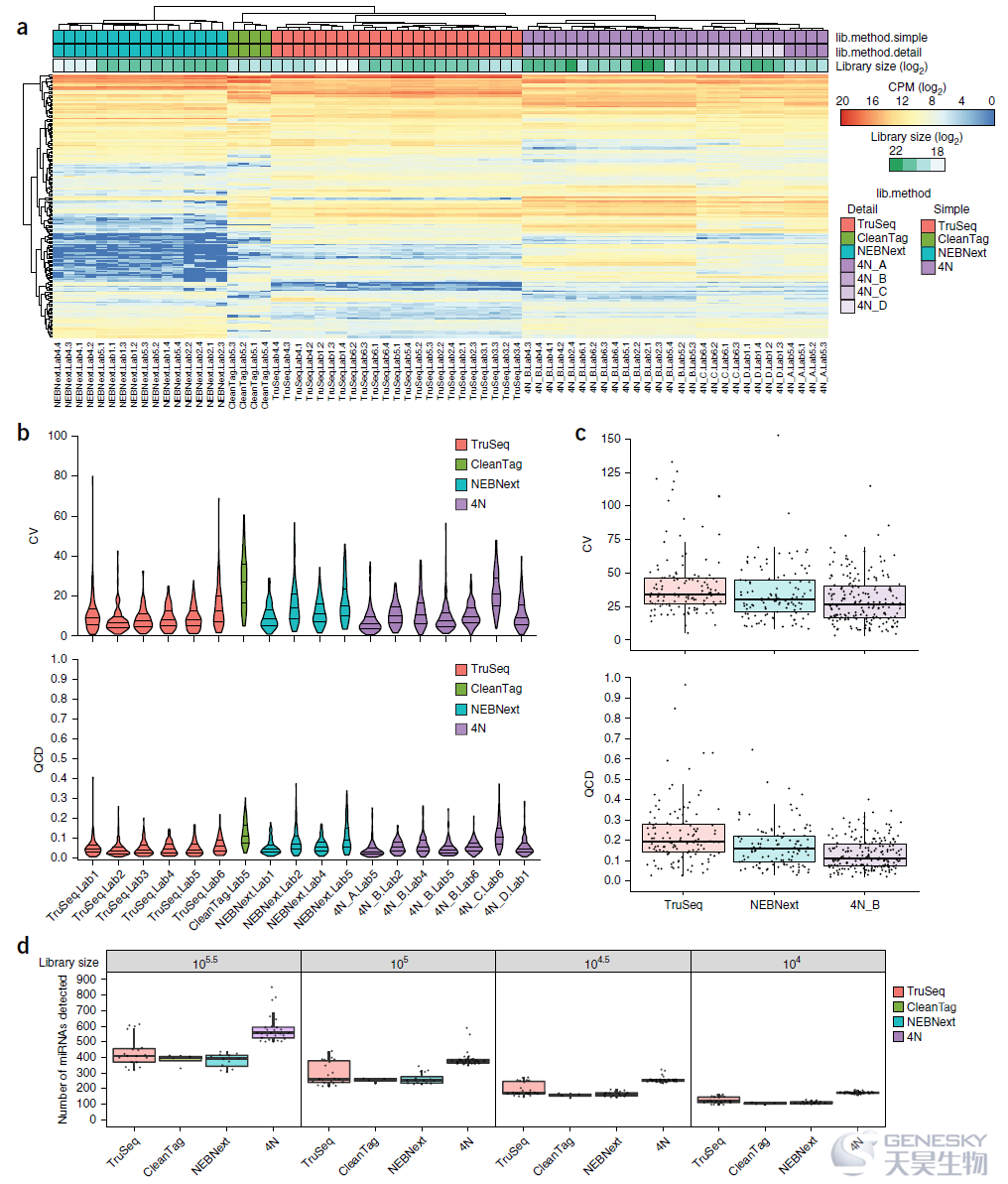
图2、等摩尔池测序结果。(a)热图显示了所有等摩尔池文库(列)中每种合成的RNA序列(行)的表达水平。表达水平代表为等摩尔池的977个长度为16-25 nt序列计算的log2 CPM(counts per million)值。行和列的分层聚类为欧氏距离的完全连锁聚类。底部标记为重复的样品名称。文库大小表示每个文库的测序深度。(b) Violin图表示了不同实验室制备的等摩尔池文库和制备方案(x轴)检测到的16-25 nt的平均CPM值(y轴)。宽度与每个位置数据点的密度成比例,水平线代表第25、50和75的百分位数。虚线显示等摩尔池中序列的预期CPM (106/977 miRNAs = 1023.5 CPM)。每个Violin图和相应的分位数线总结了n = 977个不同等摩尔池序列的平均CPM值。除了4N _ NEXTflex之外,平均CPM值是从所示每个实验室/文库制备方法的n = 4个技术复制文库中计算出来的。(c)比预期(y轴)高10倍(>10235 CPM )或低10倍(<102.35 CPM)的占比图。点和线分别表示实验室技术复制中测量的值的中位值和范围。
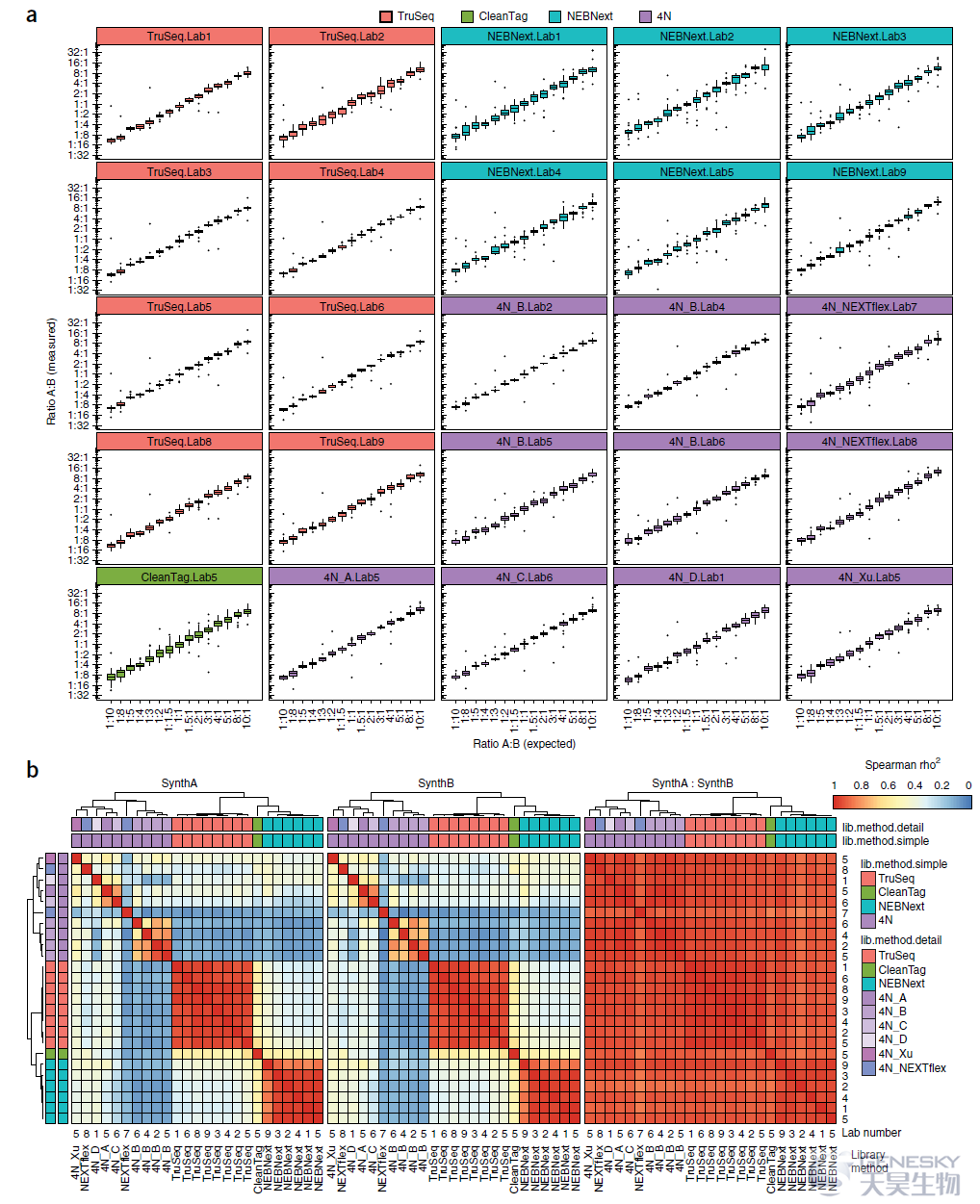
图3、小RNA-seq不同方法对样品相对表达量准确性和一致性分析。(a)为样品A和样品B亚组miRNAs预期比值(x轴)与观测比值(y轴)的关系图。(b)为样品A和样品B RNA池测序的Spearman等级相关系数热图。
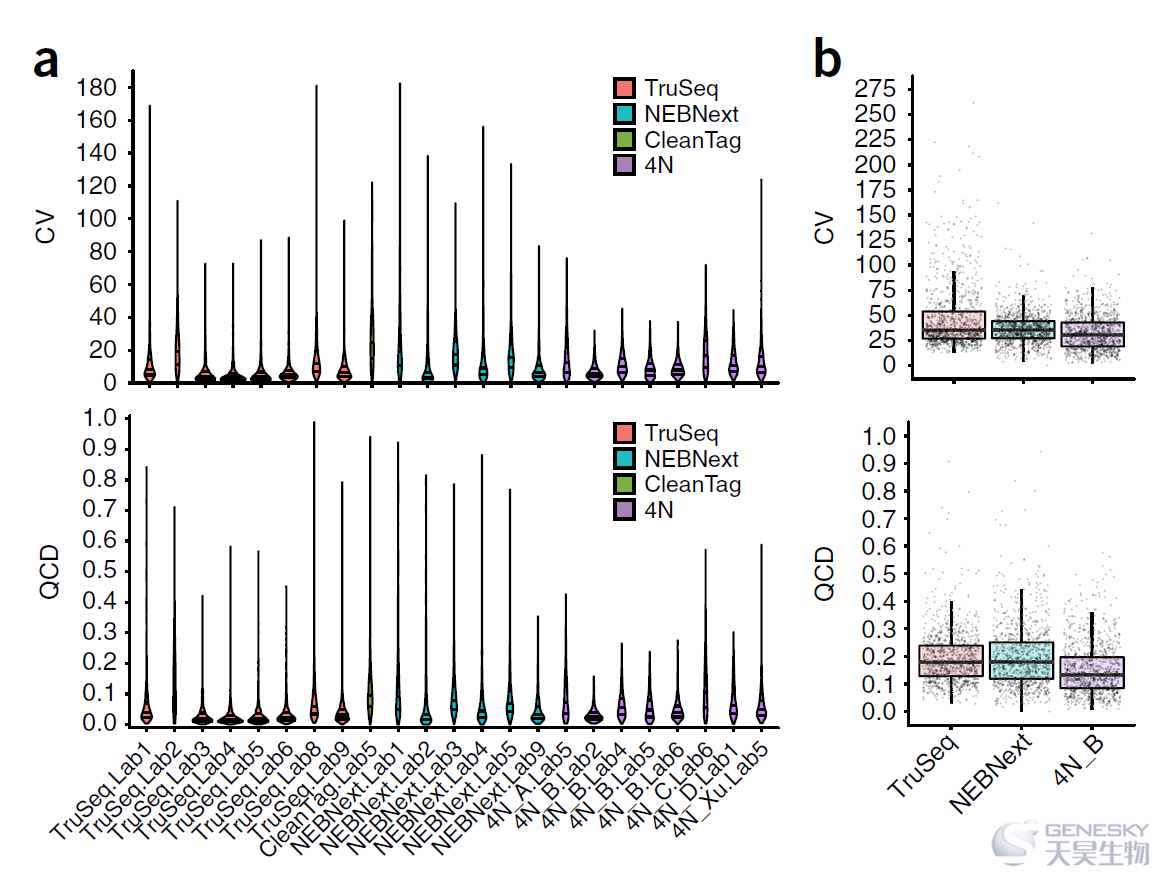
图4、实验室间以及实验室内小RNA-seq检测结果的重现性分析。(a) Violin图为从每个实验室和文库制备方法中计算出的所有等摩尔池序列定量的技术再现性分析结果,用CV (变异系数,100 x s.d./mean)和QCD (四分位数离散系数,interquartile range/average of the first and third quartile)表示,由CPM值计算获得。Violin图水平线表示第25、50和75百分位数,根据n = 977等摩尔池RNA序列的平均CPM值计算。除了TruSeq Lab1 ( n = 3 )之外,平均CPM值是从所示的每个实验室/文库制备方法的n = 4个技术重复文库中计算出来的。(b)箱线图总结了由不同实验室使用相同方案生成的等摩尔池库中测量的定量的序列特异性再现性。
图5、不同实验室使用多种文库制备方案对参考血浆RNA进行的小RNA-seq结果分析。(a)热图显示了血浆RNA文库(列)中每个序列(行)的CPM值。(b) Violin图为根据每个实验室和文库制备方法计算的血浆池文库中表达的miRNAs定量的再现性分析,以CV (顶部)和QCD (底部)百分比表示,由CPM值计算获得。(c)箱线图为使用TruSeq ( n = 6个实验室)、NEBNext ( n = 4个实验室)和4N_B ( n = 4个实验室)文库制备方案对血浆RNA池中表达的miRNAs的实验室间再现性分析结果。每个点代表针对单个miRNA跨实验室计算的CV或QCD。(d)箱线图显示不同建库方案的指定测序深度检测到的成熟miRNAs的数量。
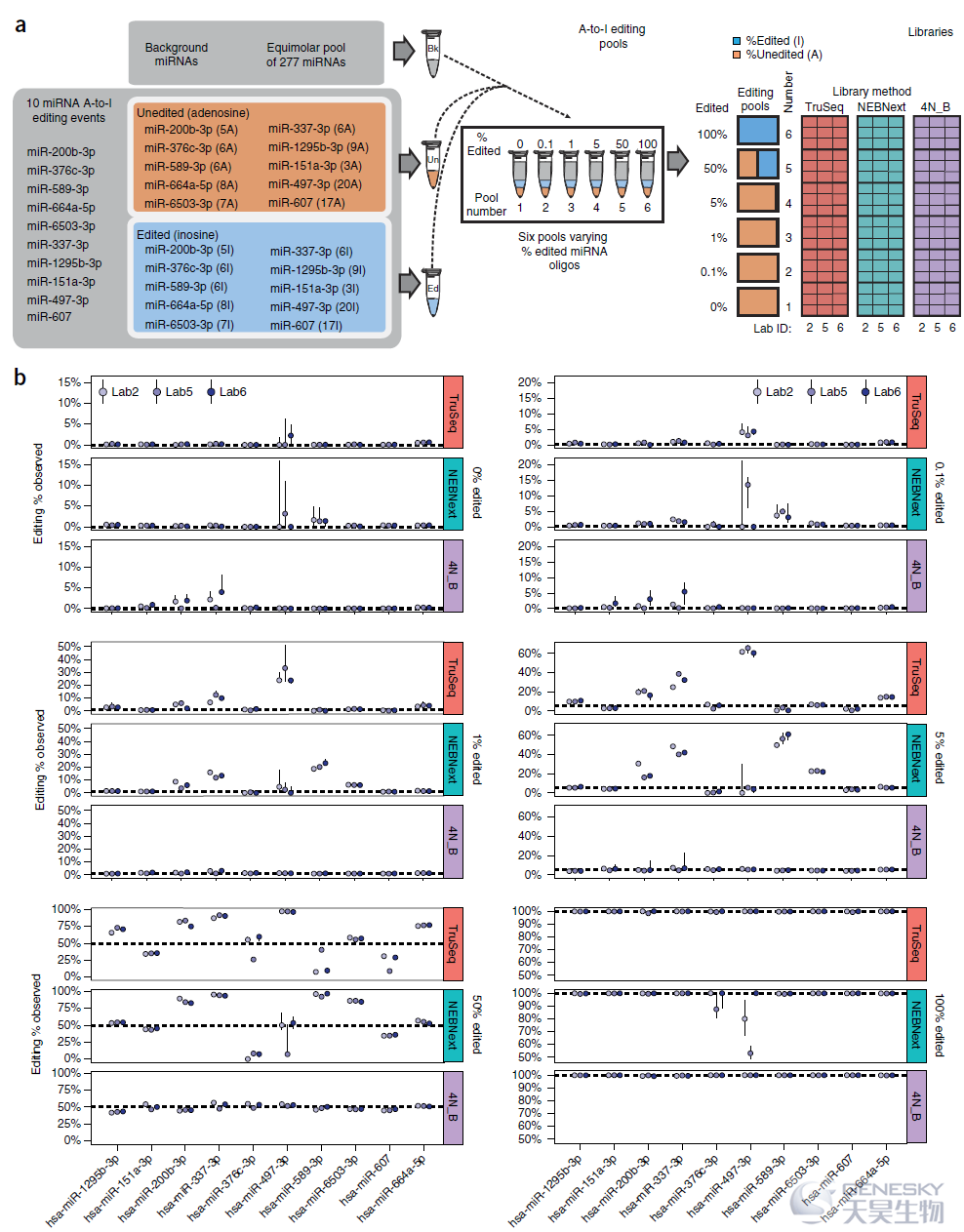
图6、测量miRNA从A到I编辑事件的不同文库构建方案性能。(a) miRNA A到I编辑实验的实验设计示意图。(b) x轴代表每种被编辑的miRNA,y轴为由A到I不同比例的RNA编辑池的观测值百分比,每个池中的预期编辑百分比显示在每组的右侧,也显示为图中的水平虚线。点和线代表三个实验室测量的百分比的中值和范围。
结论
1、小RNA-seq偏差较长RNA-seq偏差要大,尤其在检测小RNA含量较少的情况时偏差更大。
2、文库制备方案对测序偏差的影响潜在的大于实验室间差异。
3、商用试剂盒中表现最好的TruSeq对存在miRNA的检出率中位数为46%,范围在40%-55%之间。
4、所有文库制备方案对两个样本间大于1.5倍以上的差异miRNA的检出效果都较好。
5、实验室间结果的再现性是一个关键指标,结果表明常见的商业试剂盒和本实验内部4N建库制备方案,都可以将实验室间的测序CV值控制在≤20%,而且不同实验室间相对定量结果也高度一致。
关于天昊:
天昊生物,RNA-seq技术的优质服务提供商!我们通过对RNA-seq各个实验及生信分析环节不断优化,为客户提供更加准确、可靠及个性化的数据检测和分析结果,为您的科学研究保驾护航!

 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886